Chapitre 2: Algorithmes sur les graphes
Plan
Programme Officiel
|
Contenus |
Capacités attendues |
Commentaires |
|---|---|---|
|
Algorithmes sur les graphes. |
Parcourir un graphe en profondeur d’abord, en largeur d’abord. Repérer la présence d’un cycle dans un graphe. Chercher un chemin dans un graphe. |
Le parcours d’un labyrinthe et le routage dans Internet sont des exemples d’algorithme sur les graphes. L’exemple des graphes permet d’illustrer l’utilisation des classes en programmation. |

By Bumhee Park, Dae-Shik Kim, Hae-Jeong Park - http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0082873,
CC BY-SA 4.0, Link
Dans ce chapitre, nous allons voir quelques algorithmes classiques sur les graphes. Pour mémoire, un graphe est un ensemble de sommets reliés entre eux par des arêtes sans aucune contrainte sur la façon dont sont reliés les sommets par opposition aux arbres qui présente une racine, et une relation de descendance.
Présentation du module networkx
Pour travailler sur ce chapitre, nous allons utiliser la librairie networkx qui permet de facilement créer, manipuler et représenter les
graphes en Python.
Nous n'entrerons pas dans les détails de tout ce que l'on peut faire avec cette libraririe, mais nous
utiliserons la classe Graph que nous instancierons sous la variable G.
La librairie étant écrite en anglais, il faut connaitre les traductions suivantes:
- Sommet/Noeud: node
- Arête/lien: edge
- Graphe: graph
- Voisins: neighbors
import networkx as nx
import matplotlib.pyplot as plt # pour les représentations graphiques
def create_graph():
G = nx.Graph()
# Ajout des noeuds nommés
G.add_node("Paris")
G.add_node("Lyon")
G.add_node("Marseille")
G.add_node("Nice")
G.add_node("Montpellier")
G.add_node("Toulouse")
G.add_node("Rennes")
G.add_node("Nancy")
# Ajout des arêtes
G.add_edge("Paris", "Lyon")
G.add_edge("Lyon", "Marseille")
G.add_edge("Nice", "Marseille")
G.add_edge("Nice", "Lyon")
G.add_edge("Montpellier", "Marseille")
G.add_edge("Montpellier", "Toulouse")
G.add_edge("Paris", "Toulouse")
G.add_edge("Rennes", "Toulouse")
G.add_edge("Rennes", "Paris")
G.add_edge("Nancy", "Paris")
G.add_edge("Nancy", "Lyon")
return G
# création du graph
G =create_graph()
# Représenation graphique
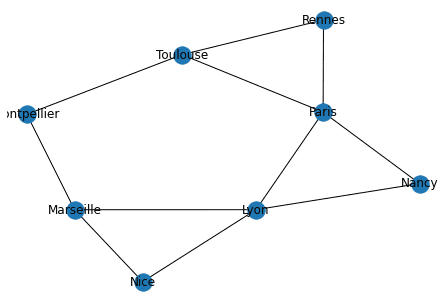
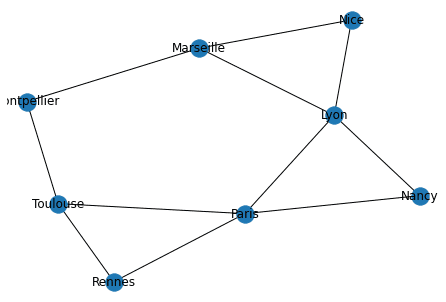
nx.draw(G, with_labels=True) # Il s'agit du graphe et non d'une carte!On peut obtenir la matrice d'adjacence représentant le graphe.
nx.to_numpy_matrix(G)matrix([[0., 1., 0., 0., 0., 1., 1., 1.],
[1., 0., 1., 1., 0., 0., 0., 1.],
[0., 1., 0., 1., 1., 0., 0., 0.],
[0., 1., 1., 0., 0., 0., 0., 0.],
[0., 0., 1., 0., 0., 1., 0., 0.],
[1., 0., 0., 0., 1., 0., 1., 0.],
[1., 0., 0., 0., 0., 1., 0., 0.],
[1., 1., 0., 0., 0., 0., 0., 0.]])Mais également sous la forme d'une liste d'adjacence comme nous l'avions vu dans le chapitre sur la structure de données graphe (ou bien d'autres formes voir doc).
nx.to_dict_of_lists(G){'Paris': ['Lyon', 'Toulouse', 'Rennes', 'Nancy'],
'Lyon': ['Paris', 'Marseille', 'Nice', 'Nancy'],
'Marseille': ['Lyon', 'Nice', 'Montpellier'],
'Nice': ['Marseille', 'Lyon'],
'Montpellier': ['Marseille', 'Toulouse'],
'Toulouse': ['Montpellier', 'Paris', 'Rennes'],
'Rennes': ['Toulouse', 'Paris'],
'Nancy': ['Paris', 'Lyon']}Parcourir un graphe
Tous comme pour les arbres, il est possible de réaliser deux types de parcours d'un arbre:
- le parcours en profondeur(Depth-First Search)
- le parcours en largeur(Breadth First Search)
Cependant, contrairement aux arbres
- il n'y a pas de racine, donc on doit choisir à partir de quel noeud on part: le noeud source.
- il peut y avoir un nombre quelconque d'arêtes, et il faut donc marquer les chemins déjà empruntés lors du parcours.
Parcours en profondeur
L'exploration d'un parcours en profondeur depuis un sommet S fonctionne comme suit. Il poursuit alors un chemin dans le graphe jusqu'à un cul-de-sac ou alors jusqu'à atteindre un sommet déjà visité. Il revient alors sur le dernier sommet où on pouvait suivre un autre chemin puis explore un autre chemin (voir vidéo ci-contre). L'exploration s'arrête quand tous les sommets depuis S ont été visités. Bref, l'exploration progresse à partir d'un sommet S en s'appelant récursivement pour chaque sommet voisin de S.
Article Wikipédia sur l'Algorithme de parcours en profondeur
Nous allons utiliser l'algorithme proposé sur l'article Wikipedia anglais:
PROCEDURE parcours_en_profondeur(G graph, s sommet)
marquer v comme visté
POUR TOUS les sommets voisins v de s FAIRE
SI v n'est pas marqué comme visité ALORS
APPELER RECURSIVEMENT parcours_en_prfondeur(G, v)Implémentation en Python
Nous allons implémenter la procédure parcours_en_profondeur proposée
précedemment.
PROCEDURE parcours_en_profondeur(G graph, s sommet)
marquer v comme visté
POUR TOUS les sommets voisins v de s FAIRE
SI v n'est pas marqué comme visité ALORS
APPELER RECURSIVEMENT parcours_en_prfondeur(G, v)Il parait préférable d'utiliser une liste d'adjacence ici puisque l'on a besoin d'accéder aux voisins fréquemment.
G = create_graph()
dg =nx.to_dict_of_lists(G)
print("Liste d'adjacence")
print("-----------------")
print(dg)
def parcours_profondeur(G, s):
# on récupère la liste des voisins
voisins = G[s]
# afficher le sommet
print(s)
# marquer le sommet s(je le supprime car visité)
# ATTENTION modification globale car passage par référence
del G[s]
# POUR TOUT sommet t voisin du sommet s
for t in voisins:
# SI t n'est pas marqué ALORS
if t in G:
parcours_profondeur(G, t)
print("\nListe des noeuds visités par notre algorithme")
print("---------------------------------------------")
print(parcours_profondeur(dg, "Nice"))
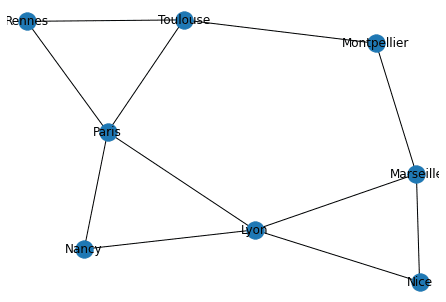
print("\nPour rappel: Forme du graphe")
print("------------------------------")
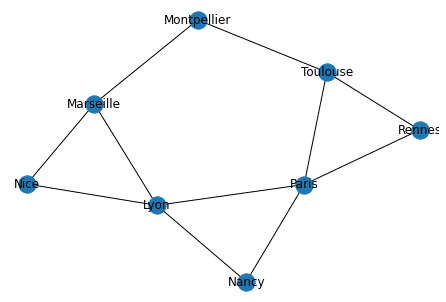
nx.draw(G, with_labels=True, pos=nx.spring_layout(G))Liste d'adjacence
-----------------
{'Paris': ['Lyon', 'Toulouse', 'Rennes', 'Nancy'], 'Lyon': ['Paris', 'Marseille', 'Nice', 'Nancy'], 'Marseille': ['Lyon', 'Nice', 'Montpellier'], 'Nice': ['Marseille', 'Lyon'], 'Montpellier': ['Marseille', 'Toulouse'], 'Toulouse': ['Montpellier', 'Paris', 'Rennes'], 'Rennes': ['Toulouse', 'Paris'], 'Nancy': ['Paris', 'Lyon']}
Liste des noeuds visités par notre algorithme
---------------------------------------------
Nice
Marseille
Lyon
Paris
Toulouse
Montpellier
Rennes
Nancy
None
Pour rappel: Forme du graphe
------------------------------Vérification avec networkx
La librairie networkx implémente cette traversée avec la méthode dfs_edges, nous allons examiner sa sortie à partir du sommet Nice pour comparer les sorties.
print("Liste des chemins suivis")
print("------------------------")
print(list(nx.dfs_edges(G, source="Nice")))
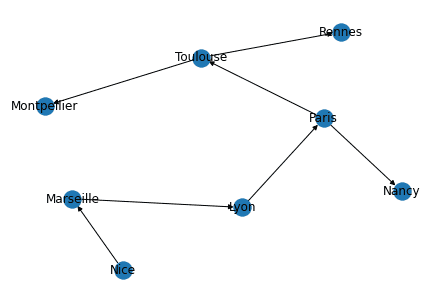
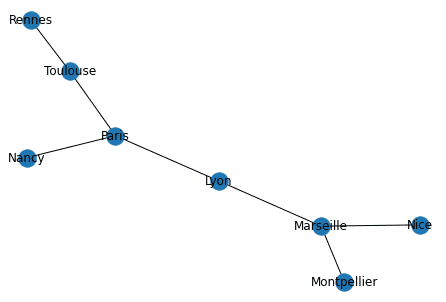
print("\nReprésentation sous forme d'arbre")
print("---------------------------------")
tree = nx.dfs_tree(G, source="Nice")
nx.draw(tree, with_labels=True, pos=nx.spring_layout(G))Liste des chemins suivis
------------------------
[('Nice', 'Marseille'), ('Marseille', 'Lyon'), ('Lyon', 'Paris'), ('Paris', 'Toulouse'), ('Toulouse', 'Montpellier'), ('Toulouse', 'Rennes'), ('Paris', 'Nancy')]
Représentation sous forme d'arbre
---------------------------------L'ordre de parcours des chemins dépend de l'ordre dans lequel les voisins sont rangés dans la liste des voisins. Cependant on observe bien que l'algorithme avance tant qu'il ne trouve pas un noeud déjà visité.
- Reproduire le graphe est l'annoter avec des flèches numérotées pour indiquer l'ordre de viste.
- Faire apparaitre les demi-tours(backtrack en anglais).
- Proposer un autre parcours en profondeur au départ de Nice.
Parcours en largeur
L'algorithme de parcours en largeur (ou BFS, pour Breadth First Search en anglais) permet le parcours d'un graphe ou d'un arbre de la manière suivante : on commence par explorer un nœud source, puis ses successeurs, puis les successeurs non explorés des successeurs, etc. L'algorithme de parcours en largeur permet de calculer les distances de tous les nœuds depuis un nœud source dans un graphe non pondéré (orienté ou non orienté).
Article Wikipédia sur l'Algorithme de parcours en largeur
Nous allons implémenter cet algorithme à l'aide d'une file:
FONCTION parcours_largeur(Graphe G, Sommet s):
f = CreerFile();
f.enfiler(s);
marquer(s);
TANT QUE la file est non vide
s = f.defiler();
afficher(s);
POUR TOUT voisin t de s dans G
SI t non marqué
f.enfiler(t);
marquer(t);Implémentation en Python
Nous allons implémenter la procédure parcours_en_largeur proposée
précedemment.
FONCTION parcours_largeur(Graphe G, Sommet s):
f = CreerFile();
f.enfiler(s);
marquer(s);
TANT QUE la file est non vide
s = f.defiler();
afficher(s);
POUR TOUT voisin t de s dans G
SI t non marqué
f.enfiler(t);
marquer(t);Encore une fois je vais utiliser une liste d'adjacence pour facilement accéder aux voisins. On utilise une
liste locale marqués pour stocker les noeuds visités.
# On importe deque pour la file
from collections import deque as file
# On repart d'un graphe tout neuf
G = create_graph()
dg =nx.to_dict_of_lists(G)
print("Liste d'adjacence")
print("-----------------")
print(dg)
# FONCTION parcours_largeur(Graphe G, Sommet s):
def parcours_largeur(G, s):
#f = CreerFile();
f = file()
#f.enfiler(s);
f.appendleft(s)
# marquer(s);
marqués = [s]
#TANT QUE la file est non vide
while f:
#s = f.defiler();
s = f.pop()
# afficher(s);
print(s)
#POUR TOUT voisin t de s dans G
for t in G[s]:
# SI t non marqué
if t not in marqués:
#f.enfiler(t);
f.appendleft(t)
#marquer(t);
# marquer le sommet s
marqués.append(t)
#node["visited"] = True
print("Liste des noeuds visités par notre algorithme")
print("---------------------------------------------")
print(parcours_largeur(dg, "Nice"))
# je ne sais pas d'ou vient ce dernier None!
print("\nPour rappel: Forme du graphe")
print("------------------------------")
nx.draw(G, with_labels=True, pos=nx.spring_layout(G))Liste d'adjacence
-----------------
{'Paris': ['Lyon', 'Toulouse', 'Rennes', 'Nancy'], 'Lyon': ['Paris', 'Marseille', 'Nice', 'Nancy'], 'Marseille': ['Lyon', 'Nice', 'Montpellier'], 'Nice': ['Marseille', 'Lyon'], 'Montpellier': ['Marseille', 'Toulouse'], 'Toulouse': ['Montpellier', 'Paris', 'Rennes'], 'Rennes': ['Toulouse', 'Paris'], 'Nancy': ['Paris', 'Lyon']}
Liste des noeuds visités par notre algorithme
---------------------------------------------
Nice
Marseille
Lyon
Montpellier
Paris
Nancy
Toulouse
Rennes
None
Pour rappel: Forme du graphe
------------------------------Vérification avec networkx
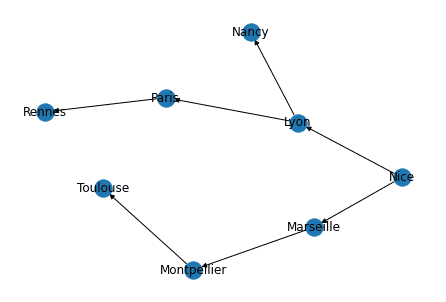
La librairie networkx implémente cette traversée avec la méthode bfs_edges, nous allons examiner sa sortie à partir du sommet Nice pour comparer les sorties.
print("Liste des chemins suivis")
print("------------------------")
print(list(nx.bfs_edges(G, source="Nice")))
print("\nReprésentation sous forme d'arbre")
print("---------------------------------")
tree = nx.bfs_tree(G, source="Nice")
nx.draw(tree, with_labels=True, pos=nx.spring_layout(G))Liste des chemins suivis
------------------------
[('Nice', 'Marseille'), ('Nice', 'Lyon'), ('Marseille', 'Montpellier'), ('Lyon', 'Paris'), ('Lyon', 'Nancy'), ('Montpellier', 'Toulouse'), ('Paris', 'Rennes')]
Représentation sous forme d'arbre
---------------------------------L'ordre de parcours des chemins dépend de l'ordre dans lequel les voisins sont visités par la méthode neighbors. Cependant on observe bien que l'algorithme explore toujours tous
les voisins d'un sommet avant d'avancer d'une profondeur supplémentaire.
- Reproduire le graphe est l'annoter avec des flèches numérotées pour indiquer l'ordre de viste.
- Proposer un autre parcours en largeur au départ de Nice.
Repérer la présence d'un cycle
- cycle
Un cycle est une suite d'arêtes consécutives (chaine simple) dont les deux sommets extrémités sont identiques.
Dans notre graphique Nice - Marseille - Lyon forme un cycle
La détection de cycle peut-être interressante par exemple en programmation concurrente dans les systèmes d'exploitation pour détecter un interblocage(deadlock) qui se produit lorsque des processus concurrents s'attendent mutuellement.
Les processus bloqués dans cet état le sont définitivement, il s'agit donc d'une situation catastrophique.
Principe
Pour détecter un cycle nous allons simplement parcourir le graphe en profondeur et vérifier qu'aucune arête pointe vers un noeud déjà visité(présence d'un backedge).
FONCTION recherche_cycle(G graph, s sommet)
marquer v comme visté
POUR TOUS les sommets voisins v de s FAIRE
SI v n'est pas marqué comme visité ALORS
APPELER RECURSIVEMENT recherche_cycle(G, v)
SINON
# On a découvert un cycle
renvoyer VRAI
# Aucun cycle découvert après parcours complet
renvoyer FAUXImplémentation
Voici le code proposé.
# On repart d'un graphe tout neuf
G = create_graph()
# Transformation en liste d'adjacence
dg = nx.to_dict_of_lists(G)
def recherche_cycle(G, s, vus=None):
# ATTENTION: Liste vide par défaut
# voir: https://www.lyceum.fr/blog/2021-04-02-comment-passer-une-liste-vide-par-defaut-a-une-fonction-en-python/
if vus is None:
vus = []
# on récupère la liste des voisins
voisins = G[s]
# marquer le sommet s
vus.append(s)
# POUR TOUT sommet t voisin du sommet s
for t in voisins:
if t in vus:
return True
# SI t n'est pas marqué ALORS
else:
recherche_cycle(G, t, vus)
return False
print("Présence d'un cycle")
print("-------------------")
print(recherche_cycle(dg, "Nice"))
# Test de la fonction à partir de tous les noeuds de départ
for node in G.nodes:
# networkx est capable de trouver des cycles
assert nx.find_cycle(G, source=node)
# on teste notre fonction maintenant
assert node, recherche_cycle(dg, node)
print("\nPour rappel: Forme du graphe")
print( "----------------------------")
nx.draw(G, with_labels=True, pos=nx.spring_layout(G))Présence d'un cycle
-------------------
True
Pour rappel: Forme du graphe
----------------------------# Nous supprimons quelques arêtes pour
# retirer les cycles et tester la fonction
def create_acyclic_graph():
G = create_graph()
G.remove_edge("Nice", "Lyon")
G.remove_edge("Nancy", "Lyon")
G.remove_edge("Paris", "Rennes")
G.remove_edge("Toulouse", "Montpellier")
return G
G = create_acyclic_graph()
# Transformation en liste d'adjacence
dg = nx.to_dict_of_lists(G)
print("Présence d'un cycle")
print("-------------------")
print(recherche_cycle(dg, "Paris"))
# Test de la fonction à partir de tous les noeuds de départ
for node in G.nodes:
try:
nx.find_cycle(G, source=node)
assert False
except nx.NetworkXNoCycle:
pass
G = create_acyclic_graph()
assert not recherche_cycle(dg, node)
print("\nPour rappel: Forme du graphe")
print( "----------------------------")
nx.draw(G, with_labels=True, pos=nx.spring_layout(G))Présence d'un cycle
-------------------
False
Pour rappel: Forme du graphe
----------------------------Recherche d'un cycle dans un graphe orienté
Le recherche d'un cycle dans un graphe orienté et plus délicate, on utilise classiquement un système de trois couleurs NOIR GRIS BLANC lors du parcours du graphe.
- BLANC: le sommet n'est pas encore traité. Au départ, tous les sommets sont BLANC.
- GRIS: le sommet est en cours de traitement (le parcours en profondeur pour ce sommet a commencé, mais pas terminé, ce qui signifie que tous les descendants (dans l'arborescence du parcours) de ce sommet ne sont pas encore traités.
- NOIR: le sommet et tous ses descendants sont traités. Si une arête est rencontrée entre le sommet actuel et un sommet GRIS, alors cette arête est l'arête arrière et il y a donc un cycle.
Chercher un chemin dans un graphe
La recherche de chemin(pathfinding), et un domaine important de recherche dans le développement de l'intelligence artificielle et de la robotique.
Plus court chemin dans un graphe non pondéré
Le plus court chemin à travers un graphe non pondéré est utilisé dans le protocole réseau RIP.
Il se base simplement sur un parcours en profondeur pour s'assurer que le nombre d'arêtes traversées est minimum.
Si le sommet est rencontré, on renverra le chemin suivi.
Pour cela on ajoute à notre algorithme un dictionnaire qui stocke la liste des prédecesseurs lors du parcours.
# On repart d'un graphe tout neuf
G = create_graph()
# On crée la liste d'adjacence
dg =nx.to_dict_of_lists(G)
# FONCTION plus court chemin(Graphe G, Sommet s, Destination d):
def plus_court_chemin(G, s, d):
# dictionnaire des prédecesseurs
prédecesseurs = {s: None}
# f = CreerFile();
f = file()
# f.enfiler(s);
f.appendleft(s)
# marquer(s);
marqués = [s]
# TANT QUE la file est non vide
while f:
# On récupère le noeud
s = f.pop()
# POUR TOUT voisin t de s dans G
for t in G[s]:
if t == d:
# Destination trouvée, on remonte le chemin
ville = s
chemin = [d]
while ville:
chemin.append(ville)
ville = prédecesseurs[ville]
# On remet dans l'ordre
chemin.reverse()
return chemin
# SI t non marqué
elif t not in marqués:
# f.enfiler(t);
f.appendleft(t)
# marquer(t);
marqués.append(t)
# màJ du dictionnaire de prédecesseurs
prédecesseurs[t] = s
# Destination non trouvée
return []
print("On teste sur tous les trajets possibles")
print("---------------------------------------")
villes = G.nodes
from itertools import combinations
for source, dest in combinations(villes, 2):
chemin = plus_court_chemin(dg, source, dest)
print(f"{source} -> {dest}: {chemin}")
print("\nSi la destination n'est pas trouvée")
print("-----------------------------------")
source, dest = "Nice", "Tokyo"
chemin = plus_court_chemin(dg, source, dest)
print(f"{source} -> {dest}: {chemin}")
print("\nPour vérification: Forme du graphe")
print("------------------------------------")
nx.draw(G, with_labels=True, pos=nx.spring_layout(G))On teste sur tous les trajets possibles
---------------------------------------
Paris -> Lyon: ['Paris', 'Lyon']
Paris -> Marseille: ['Paris', 'Lyon', 'Marseille']
Paris -> Nice: ['Paris', 'Lyon', 'Nice']
Paris -> Montpellier: ['Paris', 'Toulouse', 'Montpellier']
Paris -> Toulouse: ['Paris', 'Toulouse']
Paris -> Rennes: ['Paris', 'Rennes']
Paris -> Nancy: ['Paris', 'Nancy']
Lyon -> Marseille: ['Lyon', 'Marseille']
Lyon -> Nice: ['Lyon', 'Nice']
Lyon -> Montpellier: ['Lyon', 'Marseille', 'Montpellier']
Lyon -> Toulouse: ['Lyon', 'Paris', 'Toulouse']
Lyon -> Rennes: ['Lyon', 'Paris', 'Rennes']
Lyon -> Nancy: ['Lyon', 'Nancy']
Marseille -> Nice: ['Marseille', 'Nice']
Marseille -> Montpellier: ['Marseille', 'Montpellier']
Marseille -> Toulouse: ['Marseille', 'Montpellier', 'Toulouse']
Marseille -> Rennes: ['Marseille', 'Lyon', 'Paris', 'Rennes']
Marseille -> Nancy: ['Marseille', 'Lyon', 'Nancy']
Nice -> Montpellier: ['Nice', 'Marseille', 'Montpellier']
Nice -> Toulouse: ['Nice', 'Marseille', 'Montpellier', 'Toulouse']
Nice -> Rennes: ['Nice', 'Lyon', 'Paris', 'Rennes']
Nice -> Nancy: ['Nice', 'Lyon', 'Nancy']
Montpellier -> Toulouse: ['Montpellier', 'Toulouse']
Montpellier -> Rennes: ['Montpellier', 'Toulouse', 'Rennes']
Montpellier -> Nancy: ['Montpellier', 'Marseille', 'Lyon', 'Nancy']
Toulouse -> Rennes: ['Toulouse', 'Rennes']
Toulouse -> Nancy: ['Toulouse', 'Paris', 'Nancy']
Rennes -> Nancy: ['Rennes', 'Paris', 'Nancy']
Si la destination n'est pas trouvée
-----------------------------------
Nice -> Tokyo: []
Pour vérification: Forme du graphe
------------------------------------Plus court chemin dans un graphe pondéré
Souvent, on s'intéressera plus précisément à la recherche du plus court chemin sur des graphes pondérés, c’est-à-dire sur lesquelles on ajoute un poids à l'arête, dans notre exemple, on pourrait ajouter les temps ou distance des routes entre chaque ville.
On peut également citer le protocole réseau OSPF qui vise à optimiser les vitesses de transmission à travers les réseaux.
Il existe deux principaux algorithmes de plus court chemin, cette vidéo, vous présente l'algorithme de Dijkstra.
Un article très détaillé et illustré est disponible à cette adresse: https://perso.liris.cnrs.fr/vincent.nivoliers/lifap6/Supports/Cours/graph_traversal.html